1、首先我们将fasta文件读入python(首先选定编码形式,这里我习惯用UTF-8,中文兼容性好)。

2、这里说明一下,第一个r是表示后面的路径内容不用再转义了,最后的r表示写入。

3、然后通过循环遍历文件,注意文件是带空行的注意使用.strip(),还有文件本身是大小写都有的,小写表示的是重复序列,大写表示一般序列。所以用.upper()全部统一为大写。

4、采用"str:{0}".format()的形式结合字符串,再通过str_.count("xxx")的方式来输出特定字符的个数。
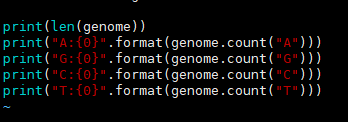
5、最后看一下效果,看来A:T和C:G的比值接近于1,所以该序列较为正常。

6、最后提一下,如果想知道其互补链的序列呢?使用自带函数str就可以啦,虽然很简单也很重要哦。

